低频变异检测(VAF<1%),在临床及科研中有着广泛的应用,例如肿瘤液体活检,微生物群中低频耐药微生物检测,以及实验室细胞系污染评估等。基于聚合酶链反应(PCR)的方法,如数字PCR、等位基因特异性PCR等可用于检测少数特定位点(1个或几个)的低频变异(0.1%VAF);NGS法可以同时对多个位点变异进行检测,然而,由于目前所有的NGS平台的平均内在误差至少为0.2%,其检测限(LOD)在1%和5%之间,基于唯一分子标签(UMI)的高深度测序法可以矫正测序中的系统误差,实现~0.1%VAF的检出限,但通常需要25,000X或更高的测序深度,即使对于中型Panel(如Guardant 36070基因Panel),每个样本测序成本也在1,000人民币以上。如此高的成本使许多研究人员、临床医生和病人无法负担。

2021年上海交通大学宋萍副教授在Nature Biomedical Engineering上发表了题为“Selective multiplexed enrichment for the detection and quantitation of low-fraction DNA variants via low-depth sequencing”的文章。其开发了一种称为多重抑制探针置换扩增技术(mBDA)的诊断方法,利用抑制探针和正向引物对不同模板杂交的热力学能差异:当模板为野生型时,抑制探针和模板的杂交更稳定,正向引物难以实现高效扩增(∆∆G=+2 kcal/mol);而当模板是突变型时,正向引物可以将抑制探针置换掉而进行高效扩增(∆∆G=-2 kcal/mol)。利用每一轮PCR过程中对野生型模板的有效抑制和对突变型模板的特异性放大,实现对超低丰度核酸突变的高灵敏检测。该技术利用计算机强大的编程技术,优化并设计了80重的引物和探针技术,检查并避免了引物二聚体(约1.9万可能性)的形成,同时将目标扩增产物比对NCBI的参考数据库,进一步优化引物设计,降低非特异性PCR产物的扩增。
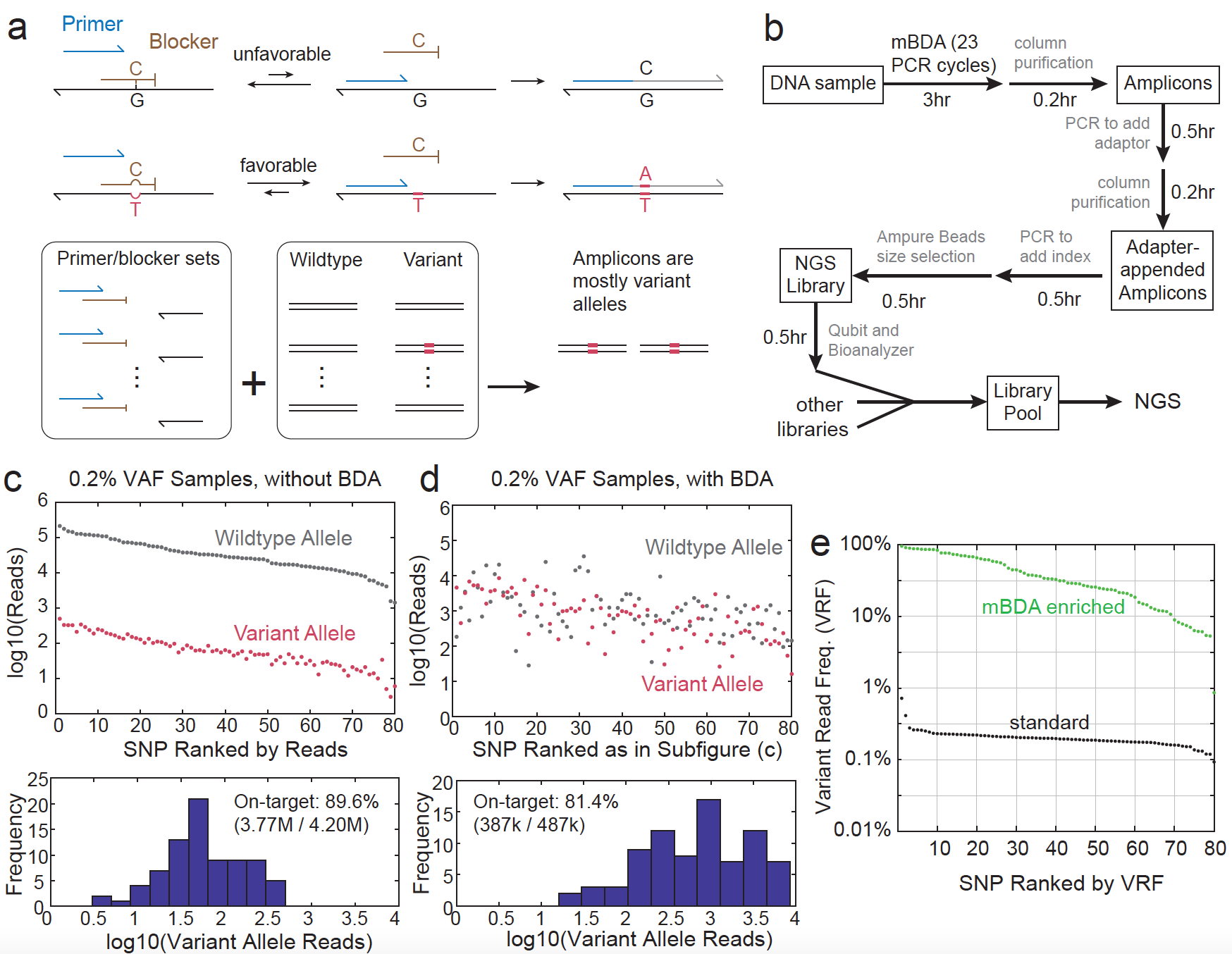
该技术的检测限最低可至0.019%,并可以将原本0.2%的突变频率经过多次PCR循环扩增到至少30%,其富集效率在150倍以上;在中靶率基本一致的基础上(mBDA 81.4% vs. 标准扩增子法 89.6%),大大降低了测序成本至少40倍;同时相比于捕获法文库构建,mBDA可将文库构建时间从24小时缩短至6h。同时该方法的抑制探针技术是目前为止最具高通量潜力的技术,一个16plex的黑色素肿瘤组织样品的panel,就可以涵盖 9个基因145个常见突变。该方法除了用于组织样品的检测,也可以实现针对cfDNA的液体活检,针对非小细胞肺癌的cfDNA的检测中,mBDA方法与UMI-based深度测序相比,其阳性一致率为78%(14/18),阴性一致率为98.6%(503/510);在定量方面,大多数变异位点在2倍区间之内(0.5-2);同时相比于深度测序平均25,000X的测序深度,mBDA-NGS仅需250X即可,在数据量上减少了近100倍。
该技术采用计算机技术和核酸探针技术相结合,优化探针设计,实现了核酸探针设计的高通量可编程,大大降低了检测成本,扩宽了其普适性,将促进更多的实验室以去中心化的模式开展检测,有望筛选出可能从靶向治疗中获益的癌症患者,从而让更多的癌症患者接受更有针对性的治疗。
论文链接:https://doi.org/10.1038/s41551-021-00713-0.

 English
English


