
开发RNA引导的核酸内切酶Cas9用于人工CRISPR分子机器代表了分子信息处理的新方向。在基于CIRPSR-Cas9系统的分子机器方向,已经发展出逻辑运算、信息存储、细胞事件记录、信号传导等功能。然而,目前对CRISPR-Cas9分子机器的操纵主要依赖于以自由扩散为基础的分子识别,这使CRISPR分子机器难以执行空间分辨的搜索或巡航等行为。
近日,王飞副教授与李茜研究员等合作开发了一种基于DNA折纸的单分子CRISPR机器,该机器可以通过自由搜索模式或局域搜索模式实现对空间分布的目标DNA分子的差异化搜寻和切割。该成果以“DNA origami-based single-molecule CRISPR machines for spatially resolved searching”为题发表在《Angewandte Chemie International Edition》期刊上。
当被封闭活性的CRISPR机器以纳米级精度在DNA折纸上的特定位点被钥匙链解锁活性后,便会开启自由搜索模式对目标DNA进行搜索(图1)。定点释放后的自由扩散带来倾向性的空间活性分布,使CRISPR机器的搜索活性随搜索距离的增大而逐渐衰减。而局域模式的构建则通过将sgRNA“拴”在折纸上实现,当Cas9蛋白结合到sgRNA上时触发局域搜索模式,sgRNA/Cas9开始搜索附近的靶标。局域搜寻模式使CIRSPR机器可及范围的目标DNA能快速被找到,而远离机器锚定位点的目标DNA则无法被找到,从而产生纳米精度的空间特异性。

图1. 基于DNA折纸的CRISPR-Cas9分子机器的运动模式。
研究者首先利用DNA折纸的空间编程能力构建靠近与远离激活位点的目标DNA。定点激活后执行自由搜索的CRISPR机器以更大的概率搜寻到近邻DNA。单分子尺度的荧光动力学观测验证了CRISPR机器搜寻成功率的空间倾向性(图2)。
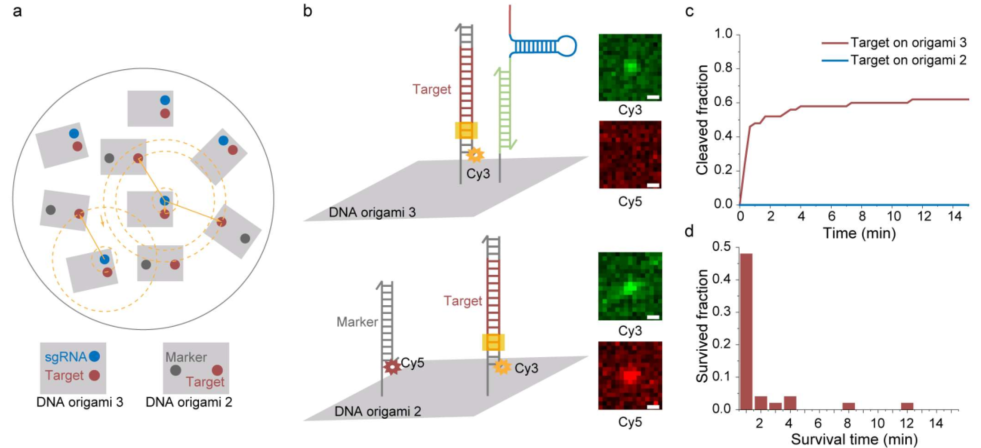
图2. 使用CRISPR分子机器通过局部研究进行空间调控的DNA切割。
局域搜索模式的单分子CRISPR机器展示了纳米精度的空间特异性。同时,纳米限域提升了CRISPR机器的搜索速度。该CRISPR分子机器为开发集成分子电路奠定了基础。该体系还可拓展至Cas12a、Cas13a等其它CRISPR体系中,可为空间分辨的高通量核酸检测的实现提供一种新策略。
上海交通大学化学化工学院化学生物学研究所李茜研究员、樊春海院士、王飞副教授为本文的共同通讯作者,博士研究生郝亚亚和助理研究员李明强为本文的共同第一作者。
原文链接:https://onlinelibrary.wiley.com/doi/full/10.1002/anie.202205460

 English
English


